
生成式互作蛋白筛选
生成式互作蛋白筛选服务介绍
1 产品概述
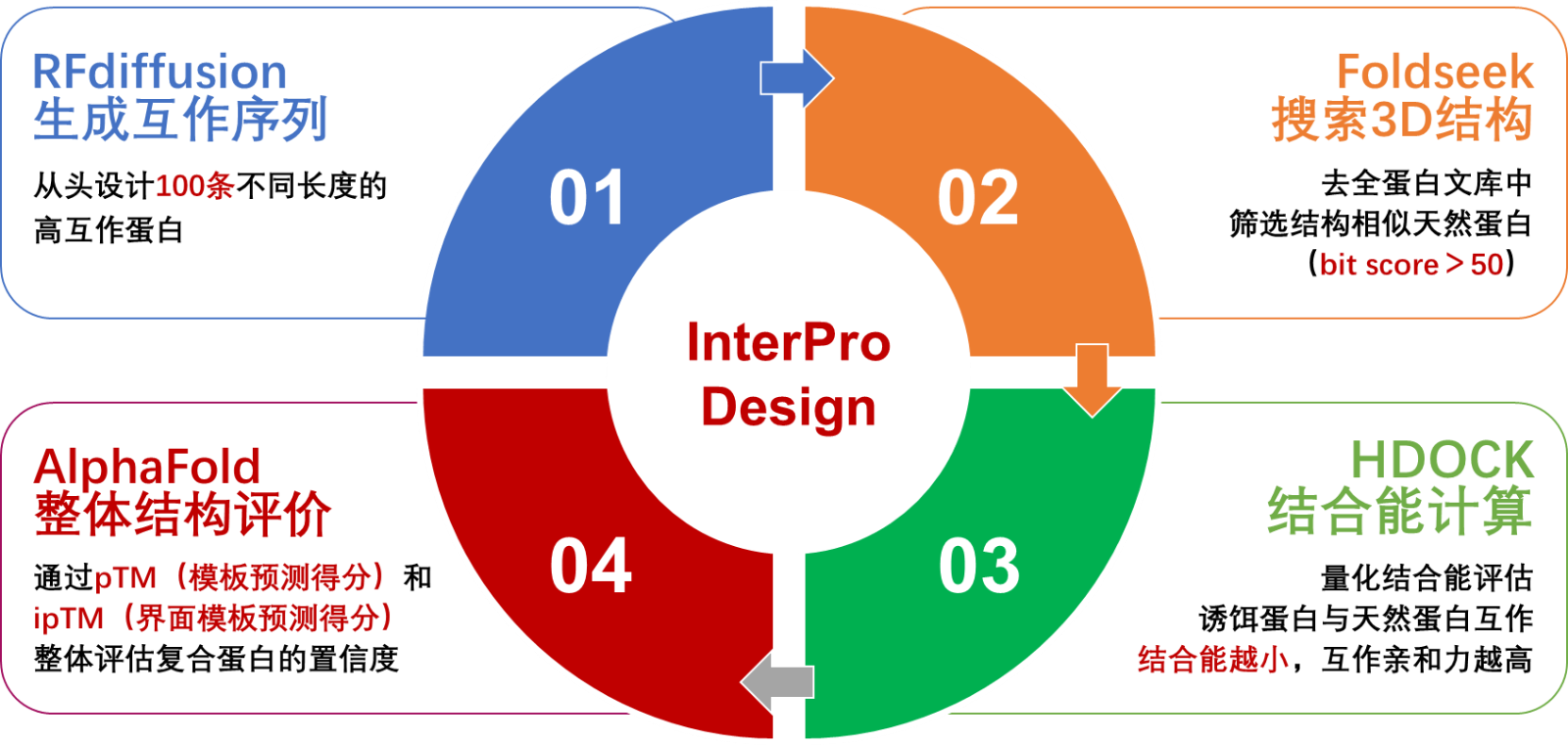
本产品旨在利用前沿的生成模型RFdiffusion从头设计人造高互作蛋白质,并结合Foldseek、HDOCK和AlphaFold 3等先进工具,对天然蛋白质进行筛选和验证。我们将这套新型计算工作流程命名为InterProDesign(IPD,Interaction Protein Design)。通过IPD能够更加高效且精准地筛选出与诱饵蛋白相互作用的天然蛋白,为进一步的实验验证提供重要参考。
2 产品筛选流程
2.1 RFdiffusion设计互作蛋白
首先,利用RFdiffusion生成模型,基于已知的诱饵蛋白结构从头设计100条可能与之高互作的蛋白质。RFdiffusion模型通过对蛋白质结合热点的推断,生成具备合理结构的蛋白质候选。
2.2 Foldseek搜索天然蛋白
将生成的100条互作蛋白分别输入Foldseek工具,针对目标物种的蛋白质三维结构数据库,寻找结构相似的天然蛋白。Foldseek通过比对蛋白质三维结构,基于结构相似性计算出bit score(仅保留bit score大于50的匹配结果),并按分数高低排序。
2.3 量化结合能评价和整体结构评估
对于上一轮Foldseek筛选出的高分天然蛋白,使用HDock工具评估它们与诱饵蛋白之间的结合能,筛选出结合能较高的天然蛋白。接着,使用AlphaFold 3进一步评估这些互作蛋白与诱饵蛋白的整体复合结构的置信度。
2.4 最终筛选天然互作蛋白
通过排列结合能和整体结构评估结果,筛选出最有可能与诱饵蛋白相互作用的天然蛋白。最终结果将为后续实验提供重点目标,显著降低了实验筛选的工作量和成本。
 科晶生物已储备物种库(部分)
科晶生物已储备物种库(部分)
3 拟交付结果
1 | 诱饵蛋白的三维结构模型,并提供建模评分 |
2 | 全部生成式结合短肽序列及PDB文件,提供与诱饵蛋白的亲和力评估 |
3 | FoldSeek比对的候选天然蛋白,结合短肽与天然蛋白的相似值 |
4 | 诱饵蛋白与Top 200 候选蛋白的HDock对接原始数据 |
5 | 诱饵蛋白与Top 20 候选蛋白的AlphaFold 3评估原始数据 |
6 | Top 200 候选蛋白的详细注释信息 |
7 | 全数据Excel表格及项目服务报告 |


